从微观角度而言,一棵参天大树不仅只有树木本身,而是由众多不同生物及其复杂相互作用共同构成的庞大生态系统。同样,乳腺癌不仅只有肿瘤细胞,也是由炎症细胞、间质细胞等众多不同细胞及其复杂相互作用共同构成的庞大生态系统。乳腺癌的生物学行为、预后和疗效不仅取决于肿瘤细胞的特征,还取决于其他细胞成分及其与肿瘤细胞的相互作用。剖析乳腺癌生态系统组成及其不同类型细胞之间的关系,对于理解乳腺癌生物学行为和提高乳腺癌精准诊断治疗水平具有重要意义。由于细胞是生命的基本单元,也是疾病发生、发展的基础。传统病理利用苏木精伊红(H&E)染色切片和光学显微镜只能对少量细胞进行描述性分析;随着数字病理被临床大规模采用,我们可以运用先进的数据和知识驱动的机器学习技术和数字图像处理技术,对高分辨数字化病理切片中数以亿计的细胞及其组织结构中多组学海量数据开展定量分析,以揭示疾病的发生、发展的趋势和规律,从而帮助我们更好地理解疾病并辅助医生制定最优的治疗策略。
2023年10月25日,智慧医疗研究院徐军教授团队与复旦大学附属肿瘤医院邵志敏教授团队合作的论文《单细胞形态和拓扑图谱揭示了乳腺癌的生态系统多样性》的论文在英国《自然》旗下《自然·通讯》杂志在线发表。智慧医疗研究院硕士生陈德品和复旦大学附属肿瘤医院赵珅、傅彤、杨竞成为本文共同第一作者。复旦大学附属肿瘤医院乳腺外科邵志敏教授、江一舟教授,病理科杨文涛教授,智慧医疗研究院徐军教授为本文共同通讯作者。在国家自然科学基金重大研究计划的支持下,由复旦大学乳腺外科与病理科以及智慧医疗研究院人工智能等多学科团队深度合作,融合机器学习、数学图论、数字图像处理等多学科知识,首创肿瘤单细胞形态与拓扑学分析算法(sc-MTOP),绘制了包含4.1亿个细胞的乳腺癌单细胞图谱,全面刻画了乳腺癌生态系统多样性,为数字病理与人工智能赋能乳腺癌精准诊疗提供了全新的视角。
首先,研究人员将sc-MTOP应用于复旦大学附属肿瘤医院637例乳腺癌(405例激素受体阳性HER2阴性、85例激素受体阳性HER2阳性、66例激素受体阴性HER2阳性、81例三阴性)数字病理切片图像进行定理分析,对约4.1亿个细胞提取单个细胞的核形态和细胞空间分布关系特征,从而可以对乳腺肿瘤生态系统进行定量分析。
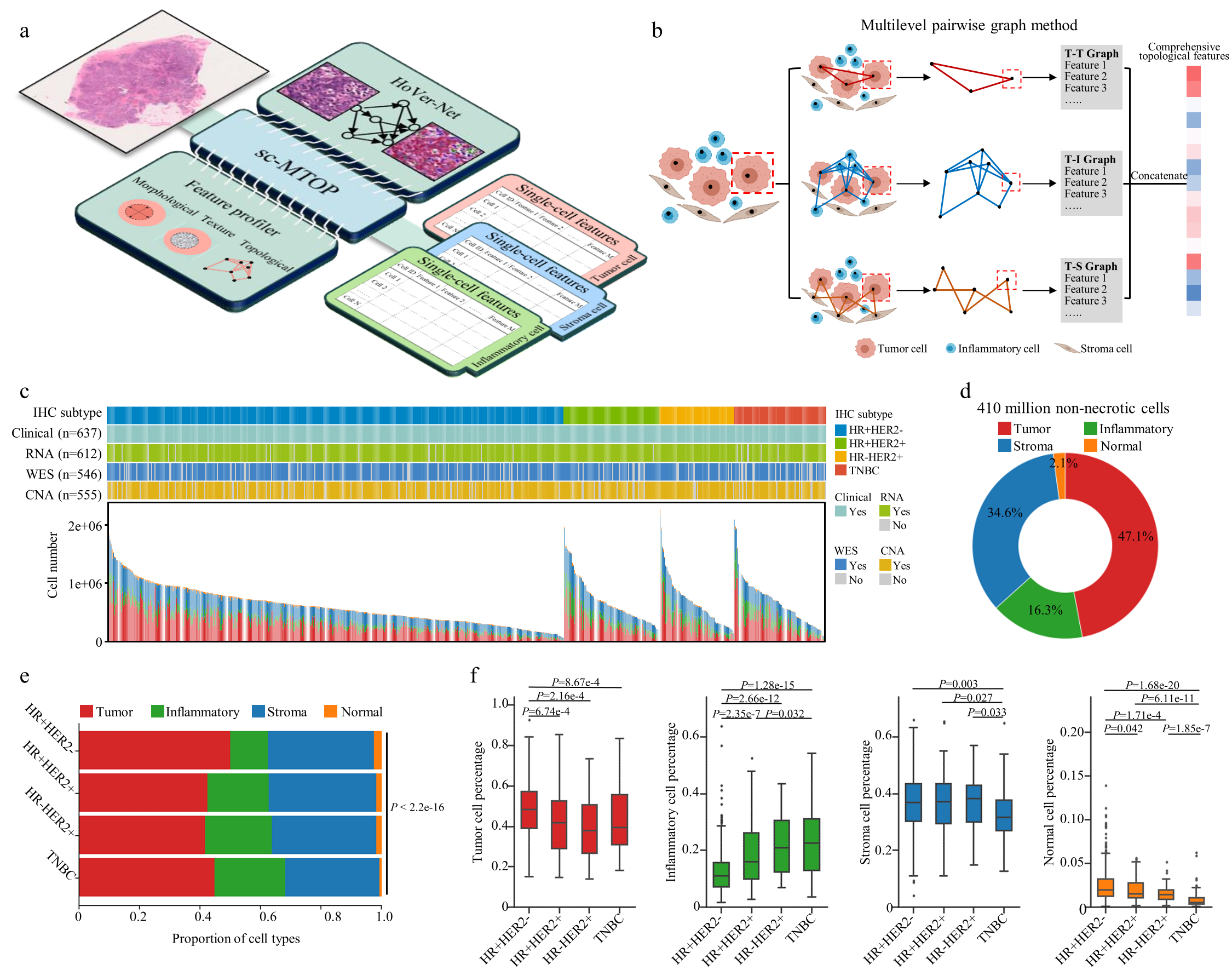
图1: sc-MTOP分析及其生成的数据集
随后,该研究构建了4.1亿个细胞的单细胞图谱,其中包括1.9亿个肿瘤细胞(47.1%)、6685万个炎症细胞(16.3%)、1.4亿个基质细胞(34.6%)、860万个正常乳腺细胞(2.1%),并分别剖析了肿瘤细胞、炎症细胞、基质细胞的表现型多样性。
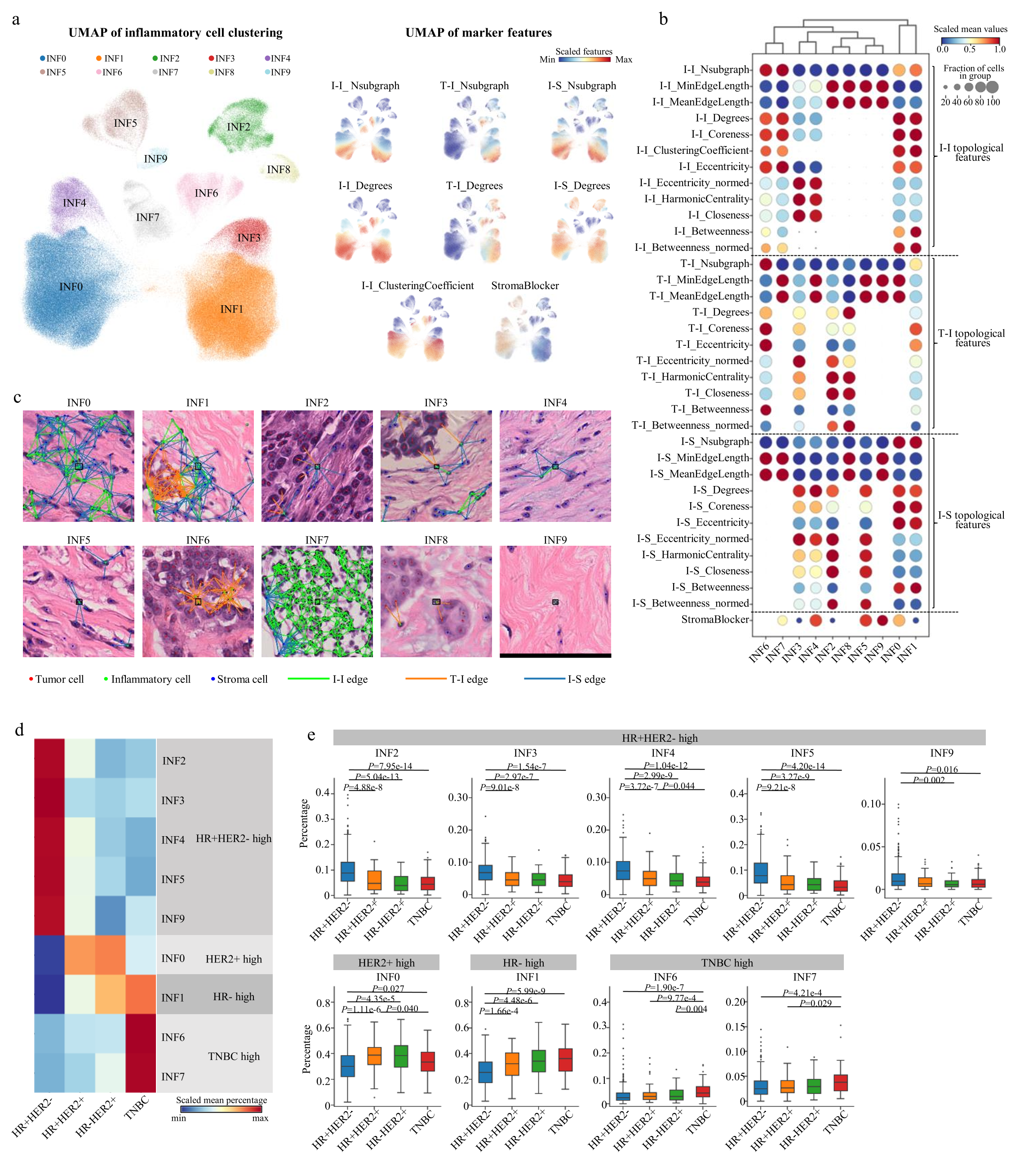
图2: 炎症细胞的单细胞图谱
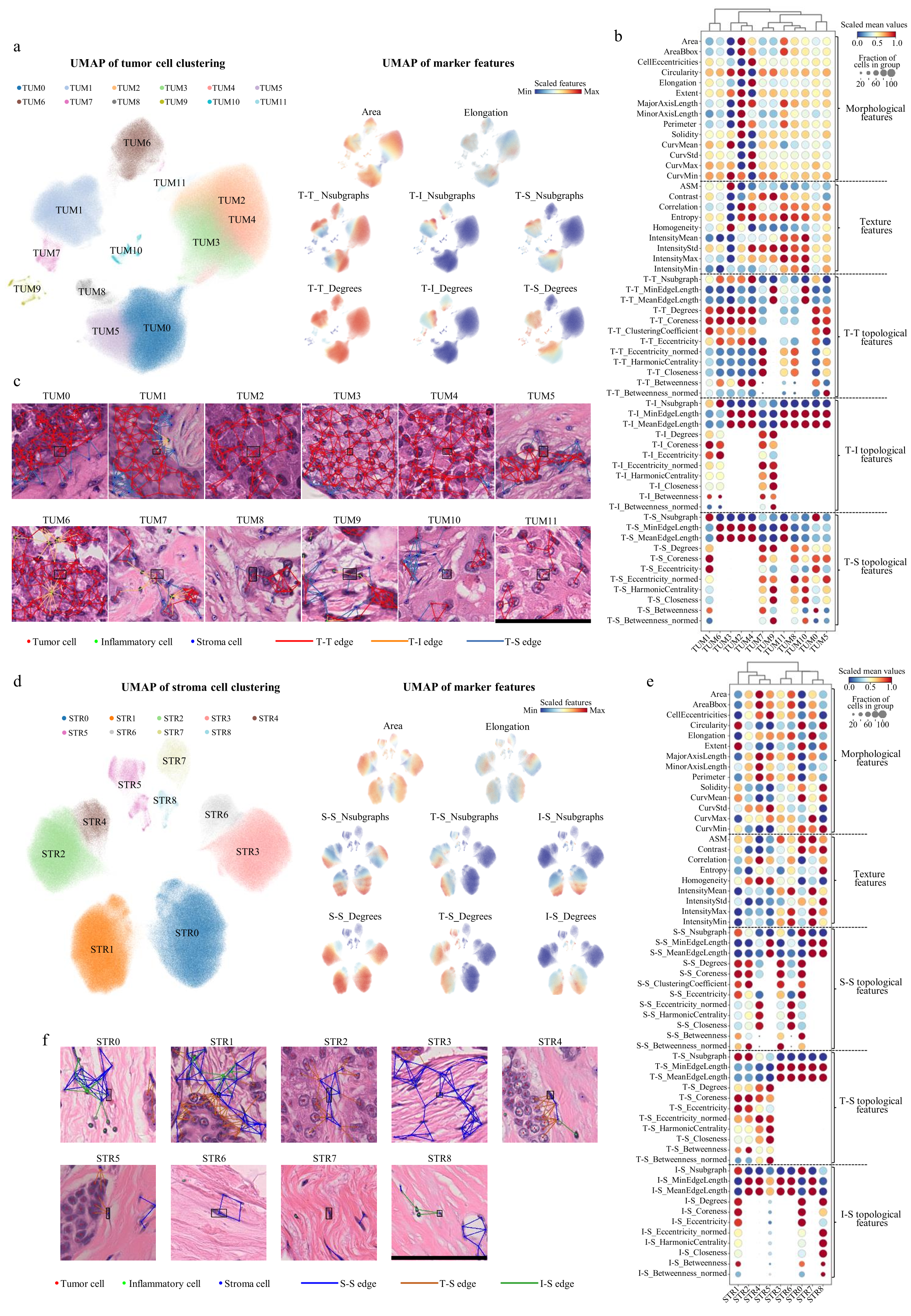
图3:肿瘤细胞和基质细胞的单细胞图谱
最后,该研究通过空间分布对代表局部多细胞结构的微生态模式与无复发生存进行分析,揭示了不同分子特征与患者无复发生存相关的四种乳腺癌生态型。
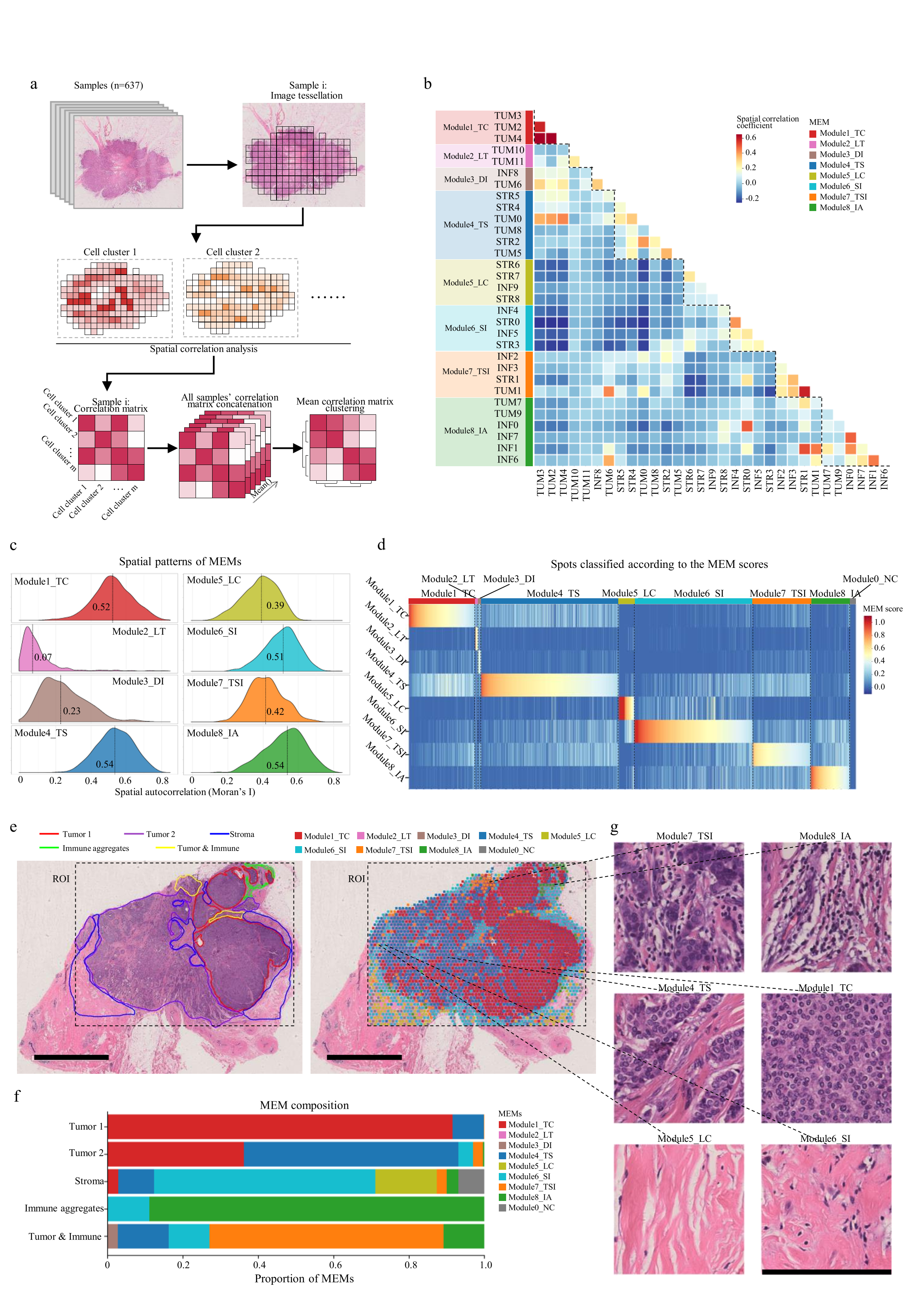
图4:肿瘤细胞和基质细胞的单细胞图谱
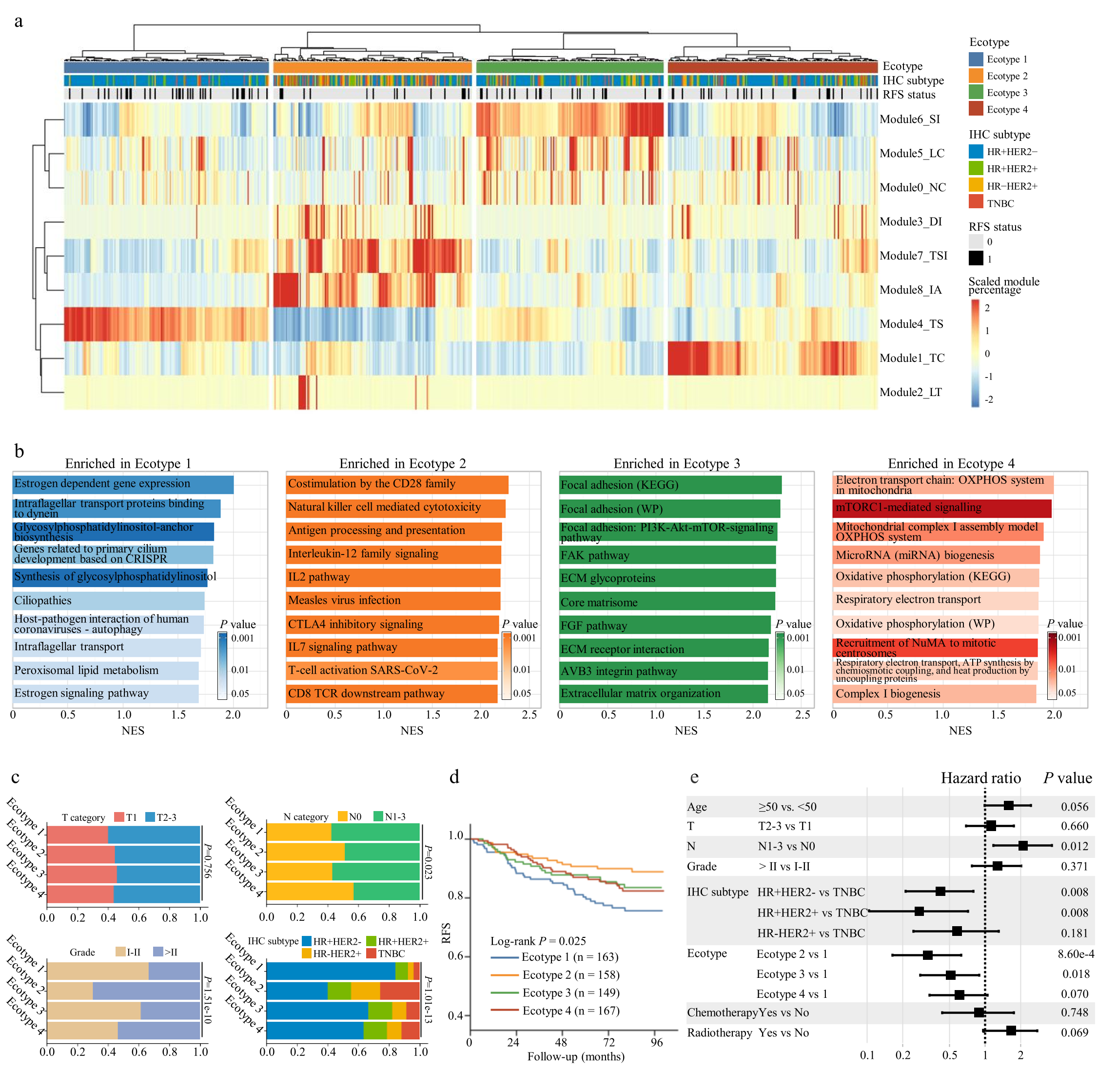
图5:不同微生态模式的乳腺癌生态型与分子特征和患者无复发生存相关
对多组学数据进一步分析,揭示了有临床意义的生态系统特征:三阴性乳腺癌局部聚集大量炎症细胞,可提示肿瘤微环境免疫激活,有利于免疫治疗效果;激素受体阳性乳腺癌肿瘤细胞核形态学异质性,可提示细胞周期通路激活和CDK抑制剂疗效。
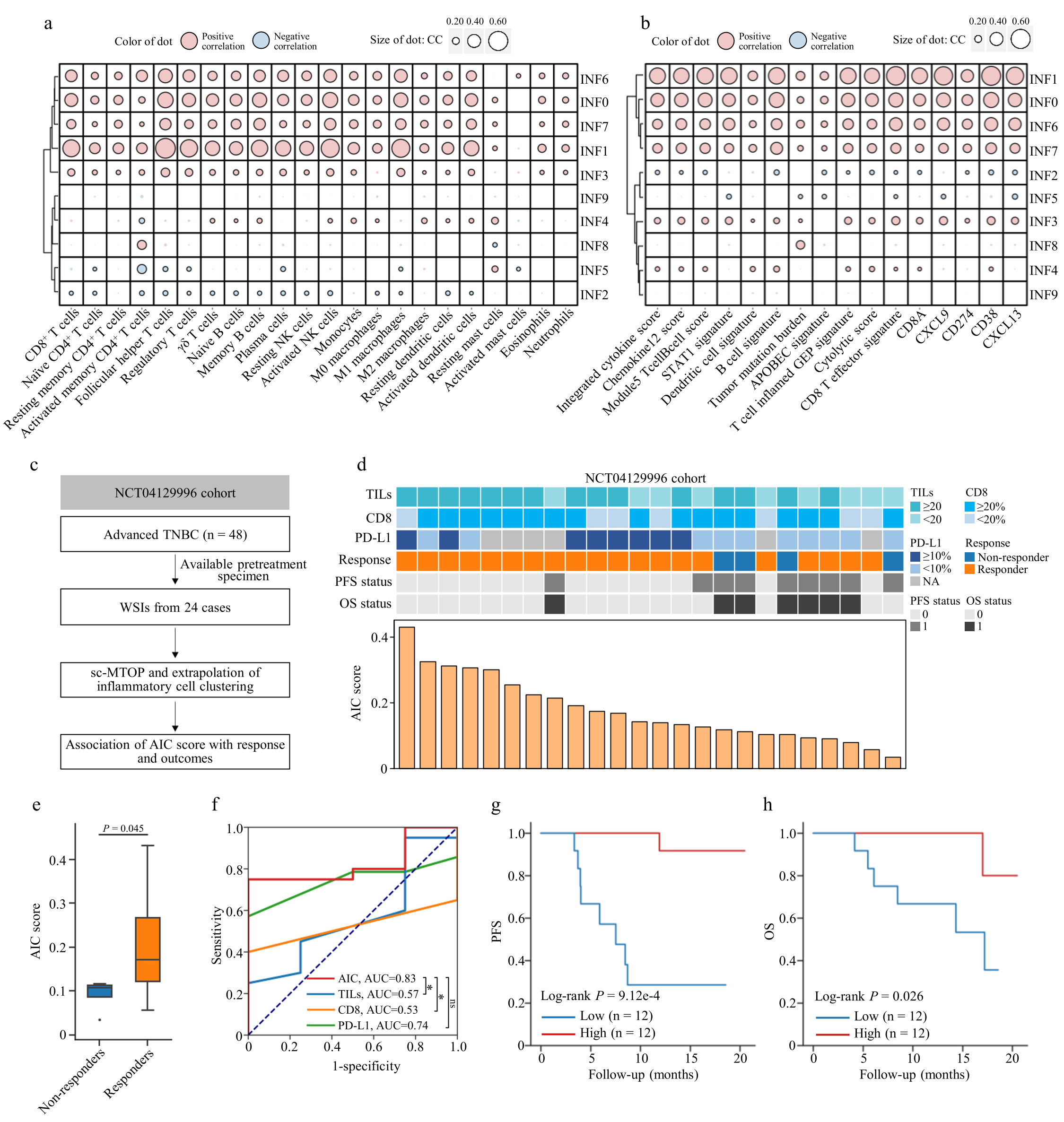
图6:三阴性乳腺癌炎症细胞局部聚集丰度可以提示免疫治疗效果
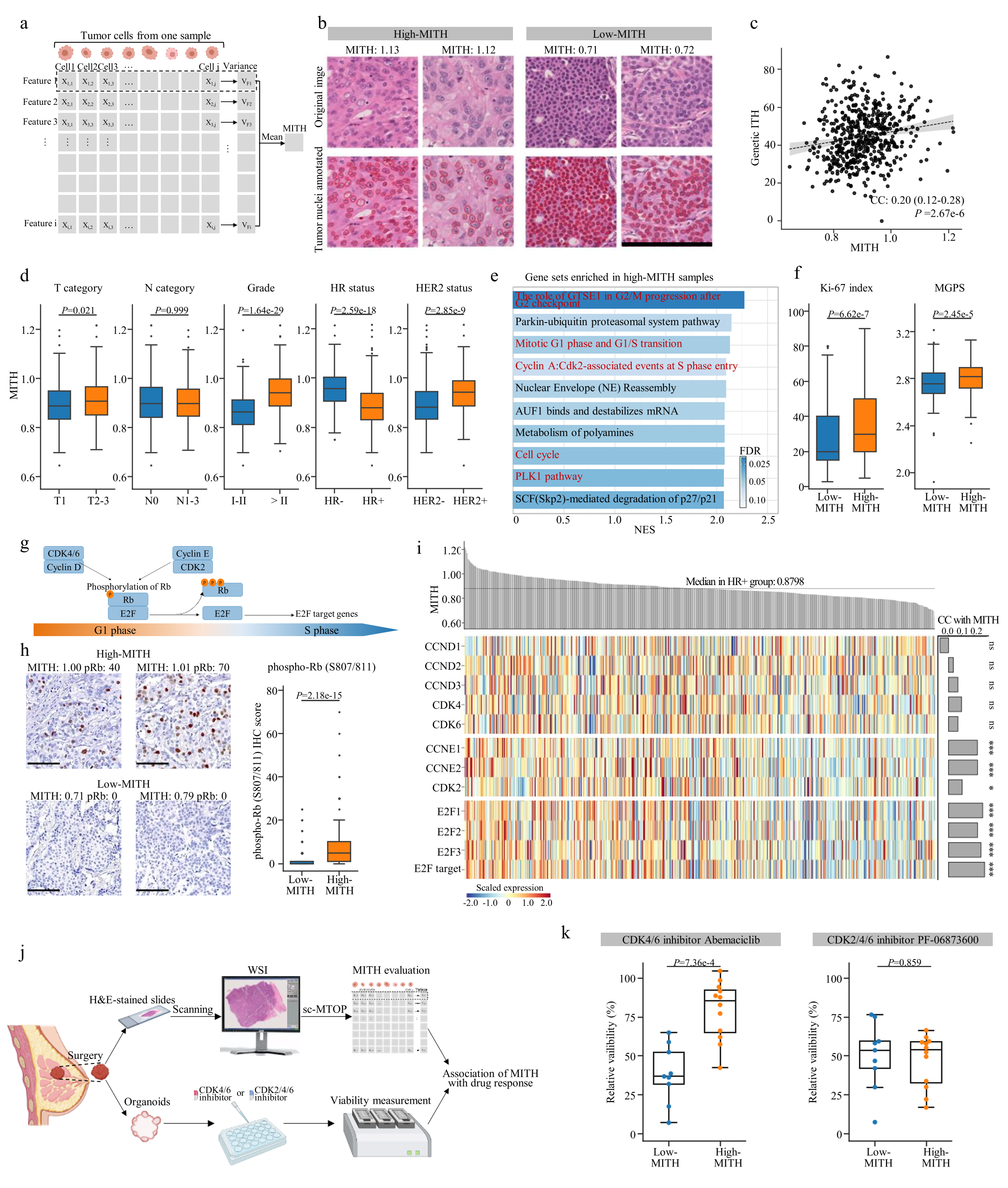
图7:激素受体阳性乳腺癌肿瘤细胞核形态学异质性可以提示细胞周期通路活性和CDK抑制剂疗效
因此,该研究结果表明,sc-MTOP算法能够对数字病理切片图像从单细胞水平剖析肿瘤生态系统,数字病理联合人工智能已经成为复旦大学附属肿瘤医院乳腺癌精准分型体系下独具特色的研究方向及重要转化工具。
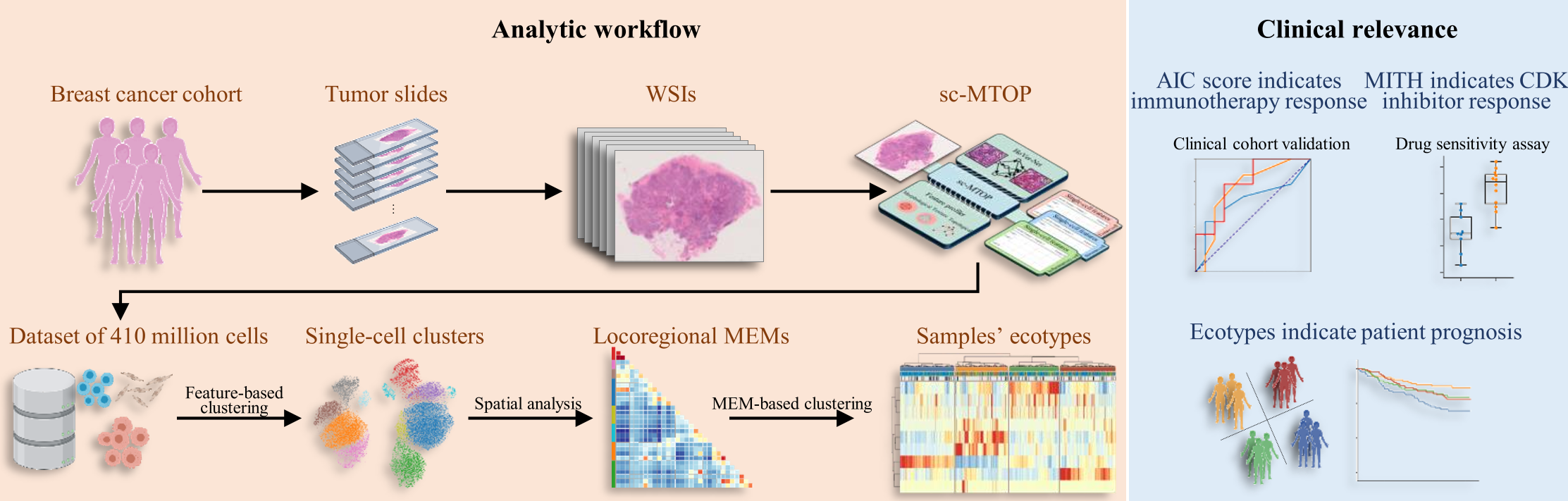
图8:研究总结
论文的引用信息如下:
Zhao, S., Chen, DP., Fu, T. et al. Single-cell morphological and topological atlas reveals the ecosystem diversity of human breast cancer. Nat Commun 14, 6796 (2023). https://doi.org/10.1038/s41467-023-42504-y