近日,南京信息工程大学多模态医学数据分析创新团队、智能医学图像计算江苏高校重点实验室(IMIC)与复旦大学附属肿瘤医院相关团队合作,在《自然》合作期刊 npj Precision Oncology 上发表。
该研究题为《基于H&E染色切片的可解释深度学习模型用于预测子宫内膜癌分子亚型》。该研究致力于基于常规H&E染色切片,整合先进图像分析和深度机器学习技术,量化提取组织形态学特征,构建能够智能区分不同分子亚型的预测模型。模型通过分析常规的病理H&E染色切片预测预测子宫内膜癌的四种主要分子亚型,在模型输入前,所有全视野切片均经过系统化的预处理流程:首先将切片切割为固定尺寸的非重叠图像块;随后通过亮度过滤排除空白或染色过度的无效区域;接着使用Vahadane方法进行染色归一化,以降低不同机构间染色差异的影响;最后,基于预训练的DeepLab-v3模型对肿瘤区域进行精确分割,确保模型学习聚焦于具有诊断意义的肿瘤形态学特征。该模型在内部交叉验证及外部独立队列中均展现出优异的分类性能与稳健的泛化能力,其综合表现优于当前多种主流弱监督学习方法。此外,通过整合Grad-CAM可视化与单细胞核特征分析,本研究增强了模型的生物学可解释性。结果不仅验证了不同分子亚型具有独特的形态学表型(如MSI-H亚型中显著的淋巴细胞浸润),还从细胞尺度定量揭示了形态特征与分子分型之间的关联。
此项技术有望显著加速临床诊断流程,辅助医生制定更精准的个体化治疗策略,为子宫内膜癌的精准诊疗提供了有力的AI支持。尽管当前模型对POLEmut等少见亚型的预测性能仍有提升空间,未来可通过扩大训练样本、引入多中心数据及算法优化进一步改进性能,推动该工具向临床实用化迈进,最终为子宫内膜癌的精准诊疗提供有效支持。
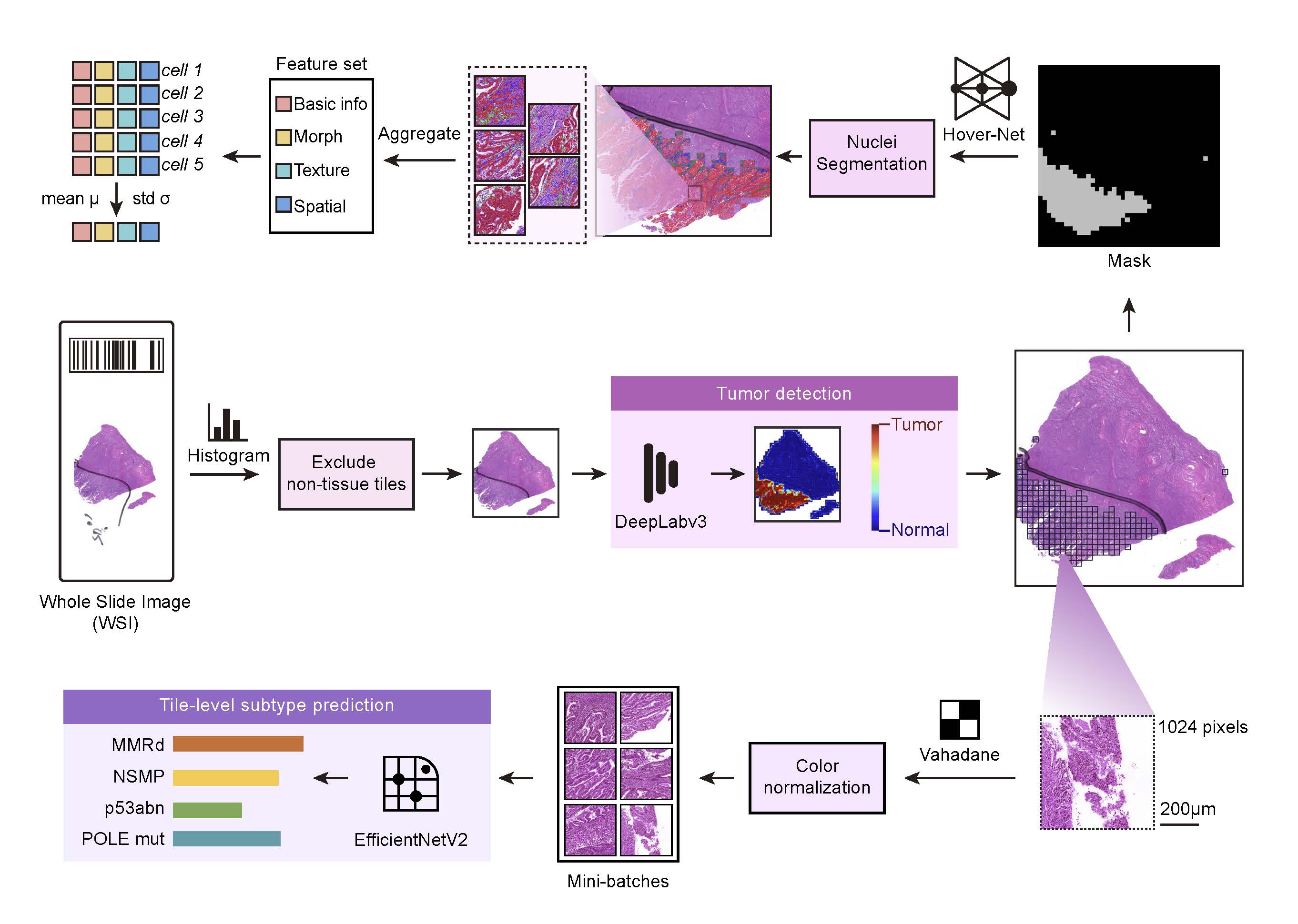
图. 本文提出的模型流程示意图
本文引用方式及原文链接:
Guo, Q., Cui, H., Zhang, Y. et al. An interpretable deep learning model for predicting endometrial cancer molecular subtypes from H&E-stained slides. npj Precis. Onc. (2026). https://doi.org/10.1038/s41698-026-01280-w