重点实验室博士生蔡程飞(导师:徐军教授)在医学人工智能领域取得进展,其关于"基于聚类和弱监督学习融合的克罗恩病治疗响应预测"研究论文被生物信息学顶尖期刊《Bioinformatics》正式录用。该成果是重点实验室与南京大学医学院附属鼓楼医院病理科(刘瑶主任、孙琦主任团队)医工交叉合作的重要体现,为克罗恩病的精准治疗提供了创新性的AI解决方案。
研究背景
克罗恩病是一种慢性炎症性胃肠道疾病,其影响可从口腔到肛门的消化道任何部位,并经常累及回肠末端和结肠,其影响几乎涉及所有年龄段的个体,超过80%的患者在40岁之前确诊。临床上,克罗恩病可能会导致肠道的狭窄、瘘管和脓肿的形成。在诊断后的10年内,约71%的患者需要接受手术以切除肠道病变。患者术后10年内症状复发率大约为40%,术后3年内内镜检查结果显示复发率为85%。克罗恩病的治疗主要包括营养支持和药物治疗,包括皮质类固醇、免疫抑制剂和生物制剂。抗肿瘤坏死因子药物,如英夫利昔单抗和阿达木单抗等,可有效诱导缓解。然而,经过一段时间的治疗后,一些患者会出现疾病复发,需要改用其他药物。因此,早期识别克罗恩病患者对特定药物的治疗响应对于疾病监测和制定最优的治疗策略至关重要。
乌斯奴单抗(Ustekinumab, UST)是一种新型单克隆抗体,该药物通过抑制白细胞介素 12/23 (IL-12/23) 信号传导来治疗克罗恩病,从而减轻炎症。它已被证明在改善临床症状、推迟手术方面有效。然而,由于缺乏可靠的组织病理学生物标志物和组织形态的复杂性,预测个体对该药物治疗的响应仍然困难。虽然最近的深度学习方法已经广泛运用了全景切片的定量分析,但大多数方法缺乏有效的机制来选择和药物治疗响应相关的区域并将图像块级特征整合到稳健的患者级预测中。因此,迫切需要一个能够捕捉局部组织学线索和全局组织背景的框架来提高预测模型性能。
研究方法
本文提出了一种新的聚类增强弱监督学习框架,该框架基于聚类和弱监督学习融合的范式,构建预测克罗恩病患者对乌斯奴单抗药物的治疗应答响应模型。该模型基于克罗恩病患者治疗前的内镜活检全景切片,研究中使用了连续的组织学样本切片进行分析,以实现克罗恩病患者对乌斯奴单抗治疗是否应答自动预测的任务。模型通过利用特征域聚类以及多实例特征融合模式,并使用患者全景切片级别的乌斯奴单抗治疗疗效是否响应作为弱监督学习模型的训练标签,从而避免了对局部区域标注的负担。
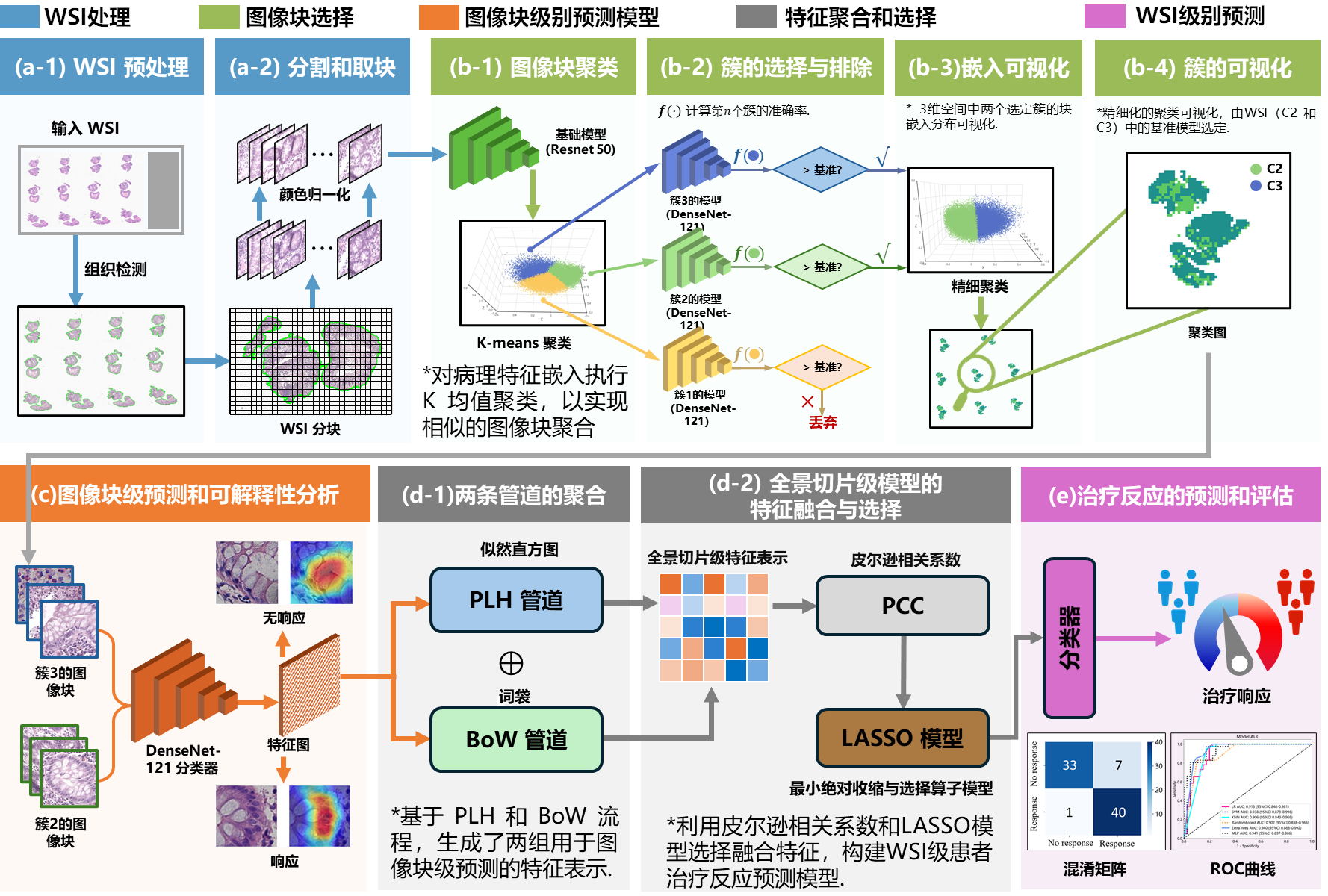
图1: 是本文研究框架概述。本文研究的预测模型框架由五个主要部分组成:(1)全景切片的预处理和标准化;(2)构建图像块筛选模型,以确定乌斯奴单抗治疗反应预测有效的图像块;(3)使用选定的图像块开发图像块级别乌斯奴单抗治疗反应预测模型;(4)融合预测的图像块结果并为每个全景切片生成特征表示;(5)建立全景切片级别的乌斯奴单抗治疗反应预测模型。
首先,本文使用预训练视觉基础模型对全景图像中的图像块进行编码,然后应用 K 均值聚类模型识别具有代表性的组织形态模式。通过基于 DenseNet 的分类器筛选出与乌斯奴单抗治疗结果相关的判别性图像块,并使用 Grad-CAM 模型对预测结果进行解释。为了汇总图像块级别的预测结果,本文采用了多示例学习方法,使用图像块似然直方图和词袋表示法构建全景图像特征。最后这些特征被用于训练分类器以进行最后环节的疗效预测。本文提出的模型在独立测试集上的实验结果表明,本文的全景切片级别模型取得了卓越的预测性能,其AUC为0.938(95% CI:0.879-0.996),灵敏度为0.951,特异性为0.825,优于目前已经发表的基线图像块级别模型。实验结果表明,本模型在测试集上表现出卓越的预测性能,能够精准评估克罗恩病患者对乌斯奴单抗的治疗反应。这些发现证实了该方法在预测生物制剂治疗响应方面的可靠性,同时模型展现出良好的可解释性和泛化能力。该预测模型具备临床应用价值,有望为医生制定个体化治疗方案提供可靠的决策支持。
实验设计
本研究采用的实验数据包括402张H&E染色组织病理全景图像,所有图像样本均源自经临床确诊的克罗恩病患者的内镜活检组织切片。研究对象的纳入标准:1)所有患者均确诊为克罗恩病;2)接受过乌斯奴单抗治疗;3)基线内镜检查提示疾病活动且SES-克罗恩病评分显著高于3分;4)随访时间至少大于24周,并完成内镜复查;参考《炎症性肠病诊治中国共识(2018北京)》,结合患者的临床表现、内镜、影像学、病理检查等综合诊断克罗恩病。排除标准:1)年龄小于18岁;2)未完成内镜复查;3)基线资料不完整;4)因非疗效相关因素更换其他药物。所有活检样本的克罗恩病患者均接受了乌斯奴单抗治疗,其中236个样本显示有乌斯奴单抗药物治疗治疗反应,166个样本无治疗反应。
实验结果
基于图像块聚类的集成模型构建中,本文随机选择了50个全景切片样本,其中29个样本具有乌斯奴单抗治疗响应的全景切片,21个样本不具有乌斯奴单抗治疗响应的全景切片。所有样本共生成了329,045个图像块作为本文中使用的 均值聚类算法的数据集。Calinski-Harabasz指数用于评估最佳聚类数。Calinski-Harabasz指数计算为聚类间方差与聚类内方差的比率。Calinski-Harabasz指数值越高,表示聚类性能越好,表明聚类分离良好且独特。其聚类结果和评估如图2所示。
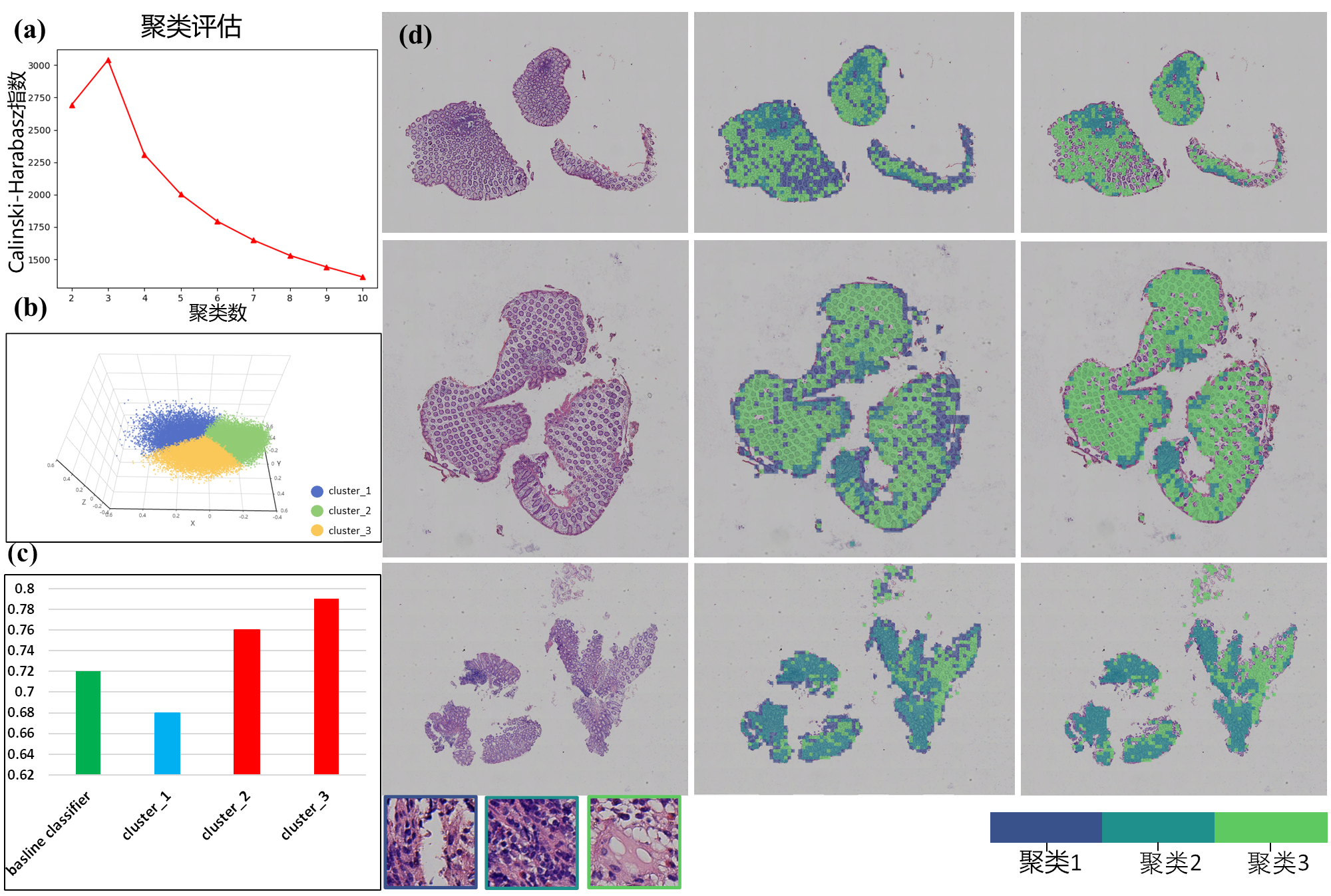
图2:图像块聚类和选择的结果
在构建通过聚类方法识别并筛选全景切片中与治疗响应高度相关的图像块后。本文构建了基于选择的图像块训练图像块级别的治疗响应预测模型,然后使用提取的病理特征训练分类器,构建全景切片级别的治疗响应预测模型,使其能够在无标注的全景切片数据上判断乌斯奴单抗对克罗恩病患者的治疗响应。其图像块级别和全景切片级别模型的预测性能如图3所示:
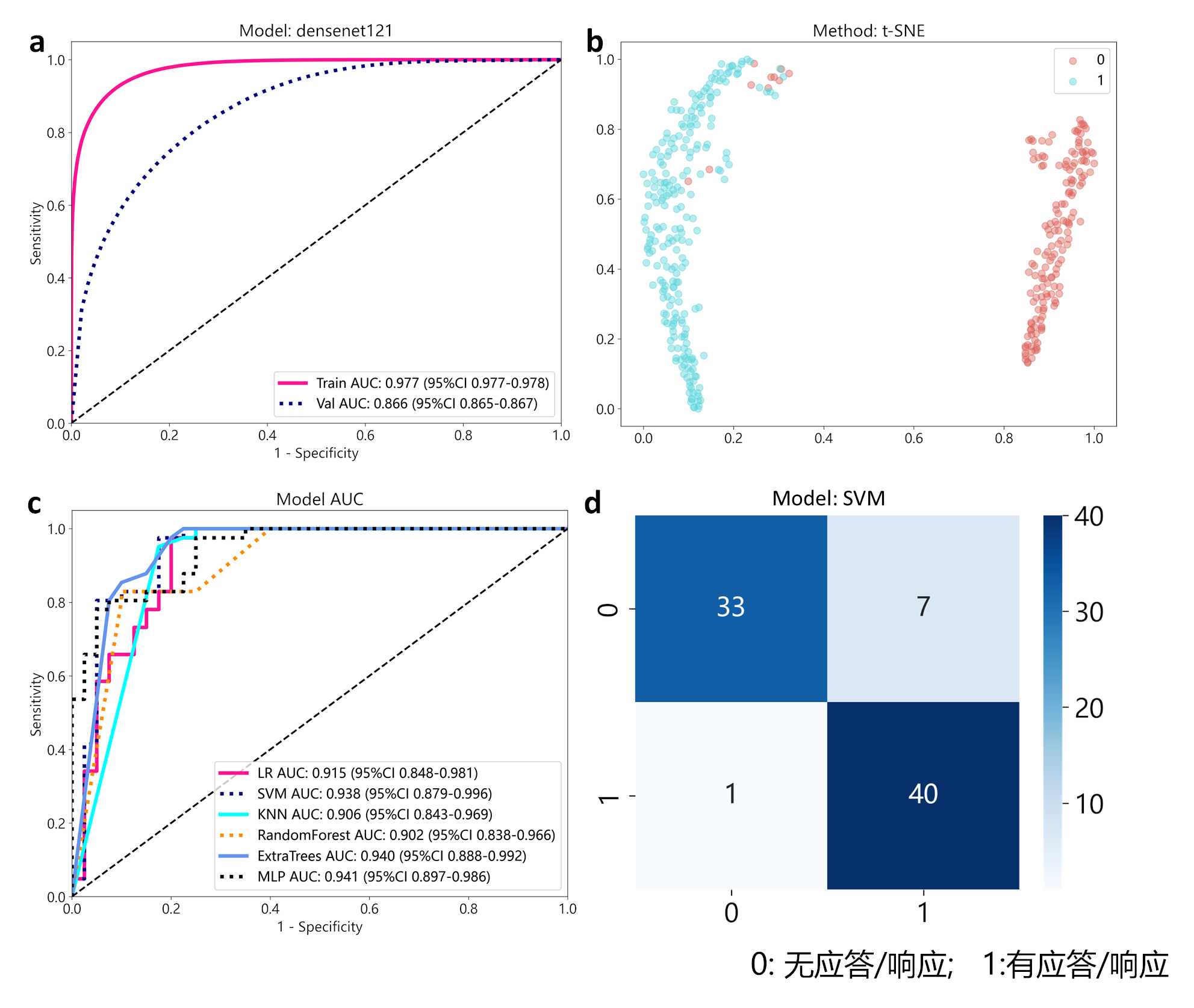
图3:图像块级模型和全景切片级模型的评估。(a) 使用 DenseNet121 模型预测图像块级的 乌斯奴单抗 响应。(b) 使用 t-SNE 可视化乌斯奴单抗响应和无响应案例在二维空间中的分布。(c) 所有分类器在全景切片级的预测结果的 AUC 值。(d) 模型在测试集中的测试结果的混淆矩阵。
结论
本研究提出了基于聚类和弱监督学习融合的预测模型,能够从克罗恩病患者的组织病理全景切片中自主学习乌斯奴单抗治疗响应的关键形态学特征。该模型具有以下创新优势:首先,采用聚类算法自动识别相似样本并筛选对分类最具贡献度的图像区域,完全避免了人工标注的需求;其次,通过选择性融合关键图像区域的判别性特征,并结合多实例学习策略实现全局诊断预测。
该预测模型基于治疗前活检全景切片数据,在测试队列中展现出卓越的乌斯奴单抗疗效预测性能。接下来,我们计划将该模型应用于前瞻性临床研究,为克罗恩病患者的个体化治疗方案制定提供决策支持。此外,该框架具有良好的可扩展性,未来可迁移至其他类似疾病的治疗响应预测任务,辅助临床医生优化治疗策略,最终改善患者临床结局。
Chengfei Cai, Ruidong Chen, Jieyu Chen, Jun Li, Caiyun Lv, Yiping Jiao, Lanqing Wu, Juan Chen, Qi Sun, Qianyun Shi, Jun Xu,Wen Tang, Yao Liu, Predicting Ustekinumab Treatment Response in Crohn’s Disease Using Pre-Treatment Biopsy Images, Bioinformatics, 2025.