智能医学图像计算江苏高校重点实验室(IMIC)博士生刘明新(导师:徐军教授)的论文“多模态表征解耦网络(MurreNet):建立组织病理学和基因组图谱之间的整体多模态相互作用模型以进行癌症患者生存期的预测”被国际医学图像计算领域顶级会议International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI 2025) 正式接收。该成果是IMIC实验室在计算病理和多模态融合领域最新的创新研究成果,为跨尺度医学数据:1)病理图像和2)基因测序数据多模态融合及癌症生存预测提供了新的解决方案。
研究背景
癌症作为全球范围内致死率较高的主要疾病之一,患者生存预后评估在临床决策中具有重要作用。准确的生存预测不仅能够帮助医生制定个性化治疗方案,还能为患者提供更科学的风险评估和随访指导。然而,肿瘤的高度异质性和复杂的病理机制,给生存预测带来了极大挑战。传统的单模态分析方法,如基于临床特征或单一分子标志物的模型,往往无法全面反映肿瘤的多维生物学特性,限制了患者预后的精确度和泛化能力。全景数字病理切片(Whole-Slide Images,WSIs)运用高分辨率成像,从肿瘤组织学形态学角度展现细胞水平和组织水平的结构及其与微环境的空间相互作用,能反映肿瘤的宏观生物学特征。与此同时,基因组学技术提供了从分子层面揭示肿瘤驱动因素的手段,包括基因突变、表达谱及信号通路的异常调控,体现肿瘤的微观分子状态。两者从不同尺度和视角揭示癌症的内在规律,具有高度互补性。
然而,在临床实践中,这两类数据通常孤立处理,难以实现有效融合。WSIs数据体量巨大、结构复杂,传统图像分析难以充分挖掘其潜在的预后信息;而基因组数据维度高且噪声大,生物学解释难度较高。如何设计科学合理的多模态融合策略,有效整合形态学与分子信息,成为提升癌症生存预测准确率的关键所在。此外,肿瘤表型与基因型之间存在复杂的相关性,捕获这种表型-基因型的耦合关系,不仅能深化对肿瘤生物学机制的理解,也有助于构建更加稳健且可解释的预测模型。当前多模态融合研究面临的主要挑战包括:不同模态间的特征分布差异显著,模态特异性与模态共享信息的区分与提取存在难度;此外,如何设计有效的交互机制,实现跨模态信息的互补与增强,也是亟需突破的瓶颈。解决上述问题,将推动基于整合病理影像与基因组数据的精准生存预测技术发展,促进临床诊断和个体化治疗策略的优化。
研究方法
为应对当前多模态生存预测面临的诸多挑战,我们提出了多模态表示解耦网络(Multimodal Representation Decoupling Network, MurreNet),其整体架构如图1所示。该网络专门针对病理图像与基因组数据之间复杂而深刻的交互关系进行建模,旨在从多模态数据中提取更加全面且层次分明的特征表示,以提升生存预测的精准性和鲁棒性。MurreNet核心理念在于强调多模态表示的解耦学习,既关注模态间的共享信息,也兼顾各自模态的特异性,进而实现信息的最大化利用与优化融合。

图1:本文所提出的多模态表示解耦网络(MurreNet)模型包括以下四个模块:(a) 病理和基因组特征的特征提取模块,(b) 多模态表征解耦模块,(c) 模态表征重建模块,(d) 通过深度整体正交融合模块进行多模态表征融合,以及用于癌症预后的综合训练正则化策略。
具体而言,本文的贡献主要体现在以下三个方面:
1) 设计了多模态表示分解模块(Multimodal Representation Decomposition,MRD),该模块通过结构化的方法系统区分模态共享(common)与模态特异(specific)表示,使得模型能够分别捕捉两类信息的内在特征,避免了传统方法中模态信息混淆带来的干扰和信息丢失,从而为后续融合提供更为清晰和解耦的表征空间。
2) 提出了深度全局正交融合模块(Deep Holistic Orthogonal Fusion,DHOF),该模块基于正交约束机制,有效整合了模态共享与模态特异信息,实现互补优势的深度融合。通过保持两类表示的正交性,DHOF不仅促进了信息的有效传递和整合,同时避免了冗余信息的重复利用,提升了模型对多模态复杂关系的表达能力。
3) 本文引入了一套综合性的损失函数组合,涵盖模态相似性、差异性与重构损失,形成多重约束机制。这一策略不仅促进了模态间共享信息的一致性,还强化了模态特异信息的独立表达,进一步保障了多模态特征的有效解耦与互补学习,为模型训练提供了坚实的理论基础和实践指导。通过在大规模癌症生存预测数据集上的广泛实验验证,我们的方法相较于当前最先进的多模态融合策略表现出显著优势,充分体现了MurreNet在捕捉复杂多模态交互、提升预测准确性和临床应用价值方面的卓越潜力。
实验结果
我们在癌症基因组图谱(The Cancer Genome Atlas, TCGA)中选取六种具有代表性的癌症类型开展了系统性实验,涵盖:膀胱尿路上皮癌(Bladder Urothelial Carcinoma, BLCA)、乳腺浸润性癌(Breast Invasive Carcinoma, BRCA)、结直肠腺癌(Colon & Rectum Adenocarcinoma, COADREAD)、肺腺癌(Lung Adenocarcinoma, LUAD)、子宫内膜癌(Uterine Corpus Endometrial Carcinoma, UCEC)以及胃腺癌(Stomach Adenocarcinoma, STAD)。在基因组信息建模方面,我们综合利用了三类核心分子数据:RNA测序(RNA-seq)、拷贝数变异(Copy Number Variation, CNV)以及基因突变状态。为了进一步提升特征的生物学解释力与结构表达,我们将这些原始数据按功能进行重组,划分为六个关键的分子子类别:蛋白激酶(protein kinases)、抑癌基因(tumor suppressor genes)、癌基因(oncogenes)、细胞分化标志物(cell differentiation markers)、转录因子(transcription),以及细胞因子与生长因子(cytokines and growth)。上述多维度的基因组子空间共同构成了模型的分子表征输入,为深入刻画病理图像与基因特征之间的跨模态关联提供了坚实的基础。实验结果如下表1所示:
表 1. 不同方法在六个公开 TCGA 数据集上的表现。“P.”表示是否使用病理图像,“G.”表示是否使用基因组图谱。最佳结果和次佳结果分别以红色和蓝色突出显示。
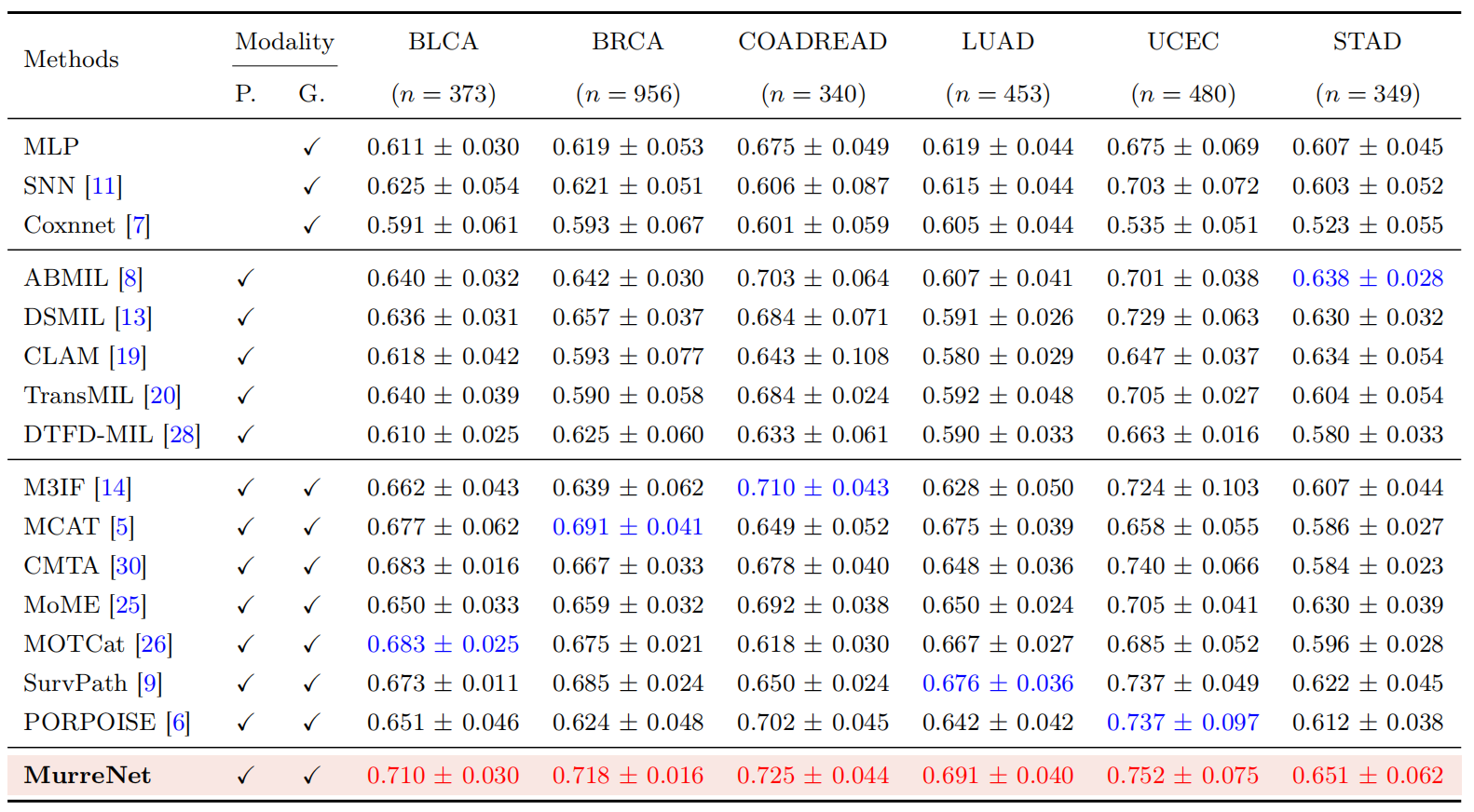
为进一步验证我们模型在生存分析任务中的实际效能,我们在六个 TCGA 癌症队列中,依据模型预测的风险评分中位数,将所有患者划分为高风险组与低风险组。随后,如图2所示,本文采用 Kaplan-Meier 生存曲线对各组患者的生存状况进行可视化展示,直观刻画生存事件的时间分布趋势。为评估组间生存差异的统计显著性,我们使用 Log-rank 检验,并以 p 值小于等于 0.05 作为显著性判据。如图2所示,在六个癌症类型中,我们提出的 MurreNet 模型均展现出显著的风险分层能力,其对应的 p 值均远低于 0.05。这一结果充分证明了模型在个体预后预测中的稳健性与泛化能力,进一步印证了其在临床风险评估和精准医疗实践中的广阔应用前景。
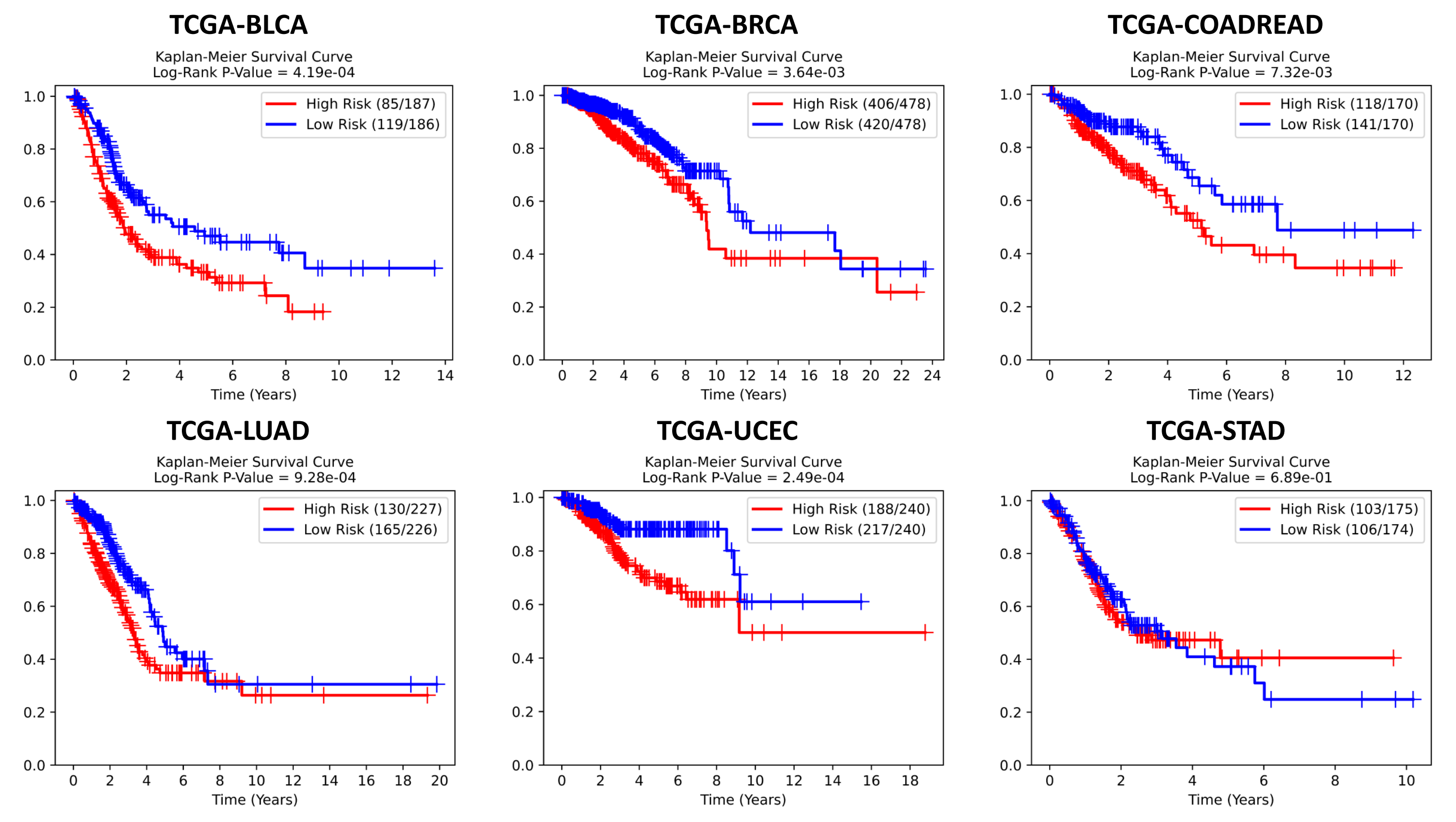
图 2. 本文所提出的多模态表示解耦网络(MurreNet)模型在六个癌症数据集上的 Kaplan-Meier 生存曲线
结论
在本研究中,我们提出了一种创新性的多模态表示解耦框架(MurreNet),旨在融合基因组数据与病理全切片图像(WSIs),以提升癌症患者的生存预测性能。该框架核心在于我们设计的 多模态表示解耦模块(MRD, Multimodal Representation Decomposition),能够系统地将多模态信息分解为模态共享特征(modality-common)与模态特有特征(modality-specific),从而实现更加精细且结构化的模态知识建模。为了增强模型训练的约束与泛化能力,我们引入了多元损失组合,包括模态相似性损失、模态差异性损失、重构损失以及生存预测损失,在保持模态间协同的同时,有效引导差异性信息的表达。进一步地,我们提出 深度全局正交融合策略(DHOF, Deep Holistic Orthogonal Fusion),用于整合共享与特有特征,实现模态间与模态内复杂关系的协同建模与深度融合。在六个 TCGA 癌症队列上开展的大量实验证明,MurreNet 在生存分析任务中显著优于当前主流方法,展现出卓越的预测准确性和稳健性,充分体现了其在精准医学领域的潜在临床应用价值。
以下是论文的引用信息:
Mingxin Liu, Chengfei Cai, Jun Li, Pengbo Xu, Jinze Li, Jiquan Ma, and Jun Xu, MurreNet: Modeling Holistic Multimodal Interactions Between Histopathology and Genomic Profiles for Survival Prediction, MICCAI 2025, September 23-27, 2025.